Eine herausragende Frage in der Biologie ist, wie Gene die Gestaltwerdung oder Morphogenese eines Organs oder eines gesamten Organismus steuern. Morphogenese ist ein vielschichtiger Prozess, der eine Vielzahl von Prozessen integriert, die auf verschiedenen Skalen wirken - von der molekularen Kontrolle der Genexpression bis zur zellulären Koordination in einem Gewebe.
Um die Morphogenese besser darstellen zu können hat ein interdisziplinäres Team aus Biologen, Physikern und Informatikern der TUM und weiterer Wissenschaftseinrichtungen im vergangenen Jahr die digitale Bildanalyse-Pipeline "PlantSeg" aufgebaut, die auf maschinellem Lernen basiert.
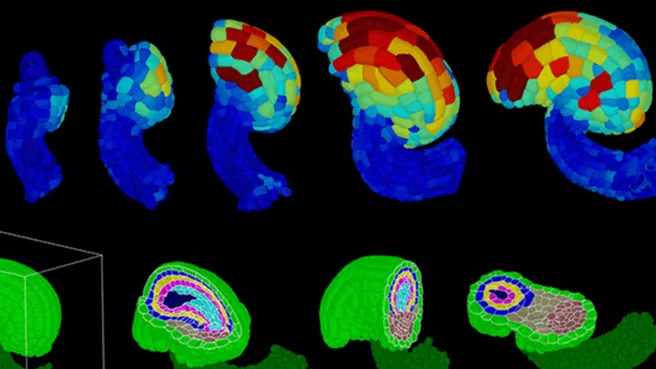
Den TU-Doktoranden Athul Vijayan und Rachele Tofanelli von der Professur für Entwicklungsbiologie der Pflanzen (geleitet von Prof. Kay Schneitz) gelang nun die erste erfolgreiche Anwendung von PlantSeg. Mithilfe modernster Mikroskopie und der PlantSeg-Pipeline konnten sie einen digitalen dreidimensionalen Atlas der Samenanlagenentwicklung in Arabidopsis thaliana erstellen.
Die Untersuchung ihrer Morphogenese stellt eine besonders schwierige Herausforderung dar, da die Samenanlage eine einzigartige gekrümmte Struktur in Kombination mit einer komplexen zellulären Organisation aufweist. Der Atlas zeichnet sich durch vollständige zelluläre Auflösung aus, auch in tiefen Gewebeschichten. Er ermöglicht eine quantitative raum-zeitliche Analyse von zellulären und Genexpressionsmustern mit Zell- und Gewebeauflösung.
So konnte zum Beispiel erstmals die ungewöhnliche Etablierung einer neuen Gewebeschicht in der Samenanlage beschrieben werden und spezifische Zellteilungsmuster in tieferen Schichten mit der Krümmung der Samenanlage in Verbindung gebracht werden. Zudem konnte die zelluläre Verteilung der Genexpression eines wichtigen regulatorischen Gens quantitativ beschrieben werden.
Diese Arbeitsergebnisse legen den Grundstein für die nächste Generation von Studien zur Morphogenese.
Video:
3D digital ovules based on deep imaging, ML-based 3D segmentation, and tissue annotation.
Publikation:
Vijayan, A., Tofanelli, R., Strauss, S., Cerrone, L., Wolny, A., Strohmeier, J., Kreshuk, A., Hamprecht, F. A., Smith, R. S., and Schneitz, K. (2021). A digital 3D reference atlas reveals cellular growth patterns shaping the Arabidopsis ovule. eLife 10, e63262.
Links:
PlantSeg Pipeline
https://elifesciences.org/articles/57613
https://github.com/hci-unihd/plant-seg
Mehr Informationen:
Die Arbeiten entstanden im Rahmen der durch die Deutsche Forschungsgemeinschaft geförderten Forschergruppe FOR 2581 „Plant Morphodynamics“. Zur Forschergruppe gehören Experten der TUM, des EMBL Heidelberg, des Max-Planck-Instituts für Züchtungsforschung in Köln, der Universität Heidelberg und des John Innes Centre in Norwich, UK. Die Mitglieder der Forschergruppe bestehen aus Biologen, Physikern und Informatikern, deren gemeinsames Interesse darin liegt zu verstehen, wie Pflanzen ihre Organe formen.
Redaktion:
Susanne Neumann
TUM School of Life Sciences
Presse- und Öffentlichkeitsarbeit
Wissenschaftlicher Kontakt:
Prof. Dr. Kay Schneitz
TUM School of Life Sciences
Professur für Entwicklungsbiologie der Pflanzen
Tel. +49 8161 71 5438
kay.schneitz(at)tum.de